- Posted by: Tommy Tran
- Tue, 28/02/2023, 8:53 (GMT+7)
- 0 Bình luận
Tập tành Sử dụng SVM trong Scikit-learn
Chắc hẳn các bạn đang tìm hiểu về Machine Learning(ML) đều biết đến một thư viện rất phổ biến cho việc lập trình các thuật toán ML trên python đó là sklearn. Bài này mình sẽ nói về cách cài đặt giải thuật SVM bằng sklearn.
Để minh họa cho SVM chúng ta dùng một tập dataset rất nổi tiếng có tên là IRIS. Tập dataset này chứa 3 lớp và 50 ví dụ cho mỗi lớp về loài cây Iris.
Load và Visualize data
import numpy as np
import itertools
from sklearn import svm, datasets
import matplotlib.pyplot as plt
from sklearn.cross_validation import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import classification_report, confusion_matrix
plt.style.use('seaborn-poster')
%matplotlib inline
iris = datasets.load_iris()
# Chỉ sử dụng hai feature đầu để visualize data
X = iris.data[:, [0, 2]]
y = iris.target
# def visualized_data():
target_names = iris.target_names
feature_names = iris.feature_names
n_class = len(set(y))
print "Number of classes:" + str(n_class)
plt.figure(figsize=(10, 8))
for i, c, s in (zip(range(n_class), ['b', 'g', 'r'], ['o', '^', '*'])):
ix = y == i
plt.scatter(X[:, 0][ix], X[:, 1][ix], color=c, marker=s, s=60, label=target_names[i])
plt.legend(loc=2, scatterpoints=1)
plt.xlabel("feature 1-" + feature_names[0])
plt.ylabel("feature 2-" + feature_names[2])
plt.show()
Train SVM
Vì mục đích chính của bài này là cách sử dụng SVM nên mình không đề cập đến các bước tiền xử lý, scale dữ liệu cũng như chia các tâp dữ liệu train, validate và test.
# def train_svm(): # khởi tạo SVM classifier clf = svm.SVC(kernel = 'linear') # Train classifier với dữ liệu clf.fit(X, y)
Kết quả train
Phần này chúng ta sẽ xem độ chính xác của model đã train thông qua confuion matrix và visualize đường bao dữ liệu dự đoán của mô hình.
iris_data = datasets.load_iris()
def plot_confusion_matrix(cm, classes, normalize=False, title='Confusion Matrix', cmap=plt.cm.Blues):
# hàm này sẽ vẽ confusion matrix
fig = plt.figure(figsize=(10,8))
plt.imshow(cm, interpolation='nearest', cmap=cmap)
plt.title(title)
plt.colorbar()
tick_marks = np.arange(len(classes))
plt.xticks(tick_marks, classes, rotation=45)
plt.yticks(tick_marks, classes)
if normalize:
cm = cm.astype('float')/cm.sum(axis=1)[:, np.newaxis]
thresh = cm.max()/2
for i, j in itertools.product(range(cm.shape[0]), range(cm.shape[1])):
plt.text(j, i, cm[i,j],
horizontalalignment="center",
color="white" if cm[i,j]>thresh else "black")
plt.tight_layout()
plt.ylabel('True label')
plt.xlabel('Predicted label')
plt.show()
def plot_desicion_boundary(X, y, clf, title = None):
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() +1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() +1
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.1),
np.arange(y_min, y_max, 0.1))
Z = clf.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.figure(figsize = (10, 8))
plt.contourf(xx, yy, Z, alpha=0.4)
plt.scatter(X[:, 0], X[:, 1], c=y, alpha=0.8)
if title is not None:
plt.title(title)
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.show()
predicted = clf.predict(X)
cm = confusion_matrix(y, predicted)
plot_confusion_matrix(cm, classes=iris.target_names, title='Confusion Matrix, without normalization')

plot_desicion_boundary(X, y, clf)

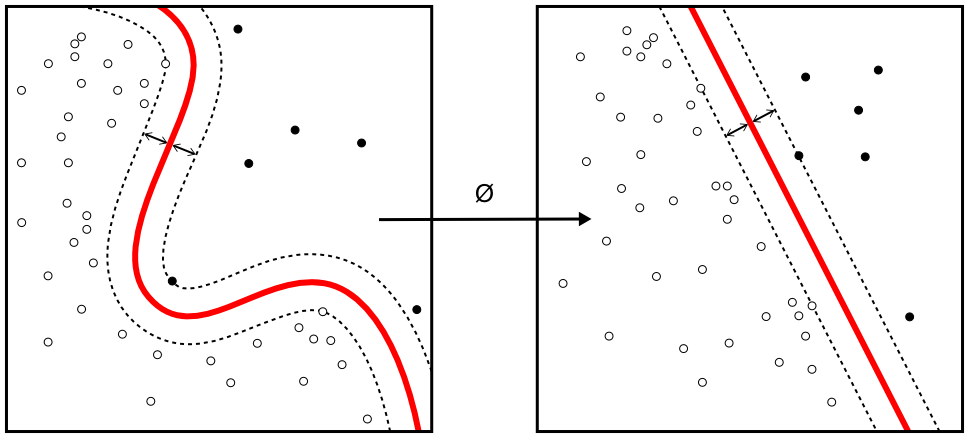
Từ hai số liệu trên chúng ta có thể thấy kết quả phân lớp của SVM với tập dũ liệu IRIS là rất tốt. Đó là kết quả với một tập dữ liễu có khả năng phân loại tuyến tính còn đối với tập dữ liệu non-linear thì sao. Chúng ta đã biết SVM có thể giải quyết các bài toán phân loại dữ liệu phi tuyến tính bằng phương pháp Kernel-Trick. Hãy xem ví dụ bên dưới.
Non-linearly separatable example
# tạo tập dữ liệu có cấu trúc phi tuyến tính X1, y1 = datasets.make_gaussian_quantiles(cov =2.,n_samples=400, n_features=2 ,n_classes=2, random_state=1) X2, y2 = datasets.make_gaussian_quantiles(mean=(3,3), cov=1.5 ,n_samples=400, n_features=2 ,n_classes=2, random_state=1) X = np.concatenate((X1,X2)) y = np.concatenate((y1, -y2+1)) n_class = 2 #Visualize dữ liệu vừa tạo plt.figure(figsize = (10,8)) for i,c,s in (zip(range(n_class), ['b', 'g'], ['o', '^'])): ix = y ==i plt.scatter(X[:, 0][ix], X[:, 1][ix], color = c, marker =s, s = 60) plt.xlabel('Feature 1') plt.ylabel('Feature 2') plt.show()

clf = svm.SVC(kernel='rbf') clf.fit(X,y) plot_desicion_boundary(X, y, clf)

Bây giờ chúng ta sẽ sử dụng SVM với hàm kernel RBF để phân loại tâp dữ liệu vừa sinh ở trên và visualize đường bao phân lớp.
Nhìn vào hình vẽ visualize đường bao chúng ta có thể thấy kết quả phân lớp là khá tốt. Khi áp dụng SVM với hàm kernel RBF để có kết quả tốt nhất ngoài các các tham số của model chúng ta còn phải tối ưu tham số C và gamma (C là tham số xác định mức độ phạt mình đã nói rõ ở bài trước, gamma là tham số của hàm RBF). Đây được gọi là các Hyperparameter các phương pháp tối ưu loại tham số này các bạn có thể tham khảo. Thông thường trong quá trình xây dựng model các tập dữ liệu validateset hay devset được dùng để tối ưu hóa hyperparameter.
Optimization C and Gamma in SVM
C và gamma là hai tham số rất quan trọng trong việc huấn luyện SVM. Chúng ta hãy cùng xem chúng ảnh hưởng như thế nào đến kết quả huấn luyện và độ chính xác của model đầu ra. Như đã trình bày ở bài trước C là tham số phạt trong bài toán soft-mirgin giúp đưa các điểm dữ liệu nằm trong khoảng hai siêu phẳng lề được phân loại đúng vào class của chúng hay giúp control misclassification. Khi C càng lớn model được huấn luyện sẽ càng fit với dữ liệu trong tập train. Điều này cũng đồng nghĩa với việc model có thể bị Overfit. Vậy chúng ta có thể rút ra được một quy luật:
+ C tăng, bias giảm, variance tăng
+ C giảm, bias tăng, variance giảm
Sau đây chúng ta sẽ lấy ví dụ cho C lần lượt có giá trị 1, 100, 10000. Nhìn vào kết quả visualize có thể thấy rằng khi C tăng biên phân lớp có xu hướng phức tạp hơn.
# C = 1 clf = svm.SVC(kernel='rbf', C=1, gamma=0.5) clf.fit(X,y) plot_desicion_boundary(X, y, clf, title= 'C = 1, gamma = 0.5') # C = 100 clf = svm.SVC(kernel='rbf', C=100, gamma=0.5) clf.fit(X,y) plot_desicion_boundary(X, y, clf, title= 'C = 100, gamma = 0.5') # C = 10000 clf = svm.SVC(kernel='rbf', C=10000, gamma=0.5) clf.fit(X,y) plot_desicion_boundary(X, y, clf, title= 'C = 10000, gamma = 0.5')



Gamma không phải là tham số của SVM mà là tham số của hàm kernel RBF. Gamma ảnh hưởng tưới model theo quy luật sau:
+ gamma tăng, bias tăng, variance giảm
+ gamma giảm, bias giảm, variance tăng
Chúng ta sẽ nhìn thấy rõ hơn qua ví dụ cho gamma nhận lần lượt 3 giá trị 0.5, 5 và 50.
# gamma = 0.5 clf = svm.SVC(kernel='rbf', gamma=0.5, C=1) clf.fit(X,y) plot_desicion_boundary(X, y, clf, title='gmma=0.5, C=1') # gamma = 5 clf = svm.SVC(kernel='rbf', gamma=5, C=1) clf.fit(X,y) plot_desicion_boundary(X, y, clf, title='gmma=0.5, C=1') # gamma = 50 clf = svm.SVC(kernel='rbf', gamma=50, C=1) clf.fit(X,y) plot_desicion_boundary(X, y, clf, title='gmma=0.5, C=1')



Trên đây là cách dùng SVM trong Sklear và một số ý cơ bản khi áp dụng SVM vào các bài toán thực tế để giúp các bạn sử dụng SVM đễ dàng và hiệu quả hơn. Chúc các bạn làm việc vui vẻ.



























Add Comment